Muster in der Wanderung von Urkeimzellen

Biologen und Mathematiker entwickeln eine neue Methode, um den Einfluss von Geweben auf die Zellbewegung zu untersuchen / Studie in „Science Advances“
Wenn ein Organismus entsteht und sich Organe entwickeln, wenn ein Tumor Metastasen bildet oder wenn bei einer Entzündung das Immunsystem aktiv wird: In all diesen Situationen wandern Zellen im Körper – und ihre Funktion wird von umliegendem Gewebe beeinflusst, mit dem sie interagieren. Die wandernden Zellen reagieren auf biochemische Signale, aber auch darauf, wie ihre Umgebung biophysikalisch beschaffen ist – also beispielsweise ob ein Körpergewebe weich oder hart ist. Erkenntnisse über solche Zusammenhänge können Wissenschaftlerinnen und Wissenschaftlern als Grundlage dienen, Ansätze für medizinische Entwicklungen zu finden.
Ein Team aus Biologen und Mathematikern der Friedrich-Alexander-Universität Erlangen-Nürnberg (FAU) und der Westfälischen Wilhelms-Universität Münster (WWU) hat jetzt eine neue Methode entwickelt, mit der sich solche Vorgänge in lebenden Organismen analysieren lassen: Die Wissenschaftler untersuchten, wie sich Urkeimzellen – Geschlechtszellen, die in ihrer Fortbewegungsart auch als Modell für andere wandernde Zellarten wie Krebszellen dienen können – unbeeinflusst von einem richtungsweisenden Lockstoff in Zebrafischembryonen verhalten. Sie entwickelten eine Software, die es ermöglicht, dreidimensionale Mikroskopieaufnahmen mehrerer Organismen zusammenzuführen, um so Muster in der Verteilung der Zellen zu finden und dadurch Gewebe zu erkennen, die die Zellwanderung beeinflussen. Mithilfe der Software bestimmten die Forscher Geweberegionen, die die Zellen entweder mieden, auf die sie mit einer Clusterbildung reagierten, oder in denen sie ihre normale Verteilung beibehielten. Auf diese Weise identifizierten sie auch eine physikalische Barriere im Bereich der Längsachse des Organismus, an der die Zellen ihre Bewegungsrichtung ändern. „Wir erwarten, dass unser experimenteller Ansatz und die neu entwickelten Werkzeuge in der Forschung zu Entwicklungsbiologie, Zellbiologie und Biomedizin von großem Nutzen sein werden“, betont Prof. Dr. Erez Raz, Zellbiologe am Zentrum für Molekularbiologie der Entzündung (ZMBE) der WWU und Leiter des Projekts. Die Studie ist in der Fachzeitschrift „Science Advances“ erschienen.
Details zu Methoden und Ergebnissen
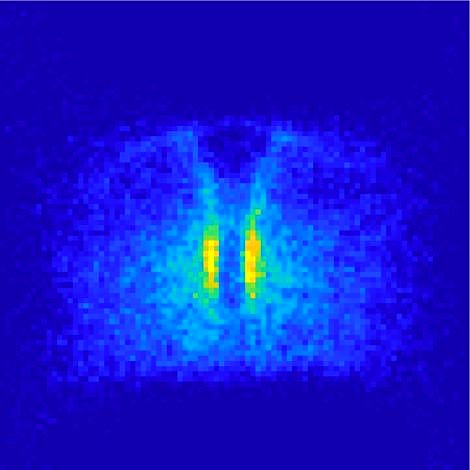
Für ihre Untersuchungen nutzten die Wissenschaftler die Eigenschaften von Urkeimzellen in Zebrafischembryonen. Urkeimzellen sind die Vorläufer von Spermien und Eizellen und wandern in der Entwicklung vieler Organismen zu dem Ort, an dem sich die Geschlechtsorgane ausbilden. Normalerweise werden sie dabei von Chemokinen geleitet. Dies sind Lockstoffe, die von umliegenden Zellen gebildet werden und Signalwege in Gang setzen, indem sie an Rezeptoren auf den Urkeimzellen binden. Durch eine genetische Veränderung der Zellen deaktivierten die Wissenschaftler den Chemokinrezeptor Cxcr4b, so dass die Zellen weiter beweglich blieben, aber nicht mehr zielgerichtet wanderten. „Unsere Idee war, dass uns die Verteilung der Zellen im Organismus – wenn sie nicht durch Lockstoffe gesteuert werden – Hinweise darauf geben kann, welche Gewebe die Zellwanderung beeinflussen, und dass wir dann deren Eigenschaften analysieren können“, erklärt Łukasz Truszkowski, einer der drei Erstautoren der Studie.
„Um über die räumliche Verteilung der migrierenden Urkeimzellen statistisch aussagekräftige Daten zu erhalten, müssen wir uns mehrere Hundert Zebrafischembryonen anschauen – denn ein einzelner Fisch hat in dem Entwicklungsstadium, in dem die Zellen aktiv wandern, durchschnittlich nur etwa 20 Urkeimzellen“, erläutert Sargon Groß-Thebing, ebenfalls Erstautor und wie sein Kollege Biologiedoktorand im Graduiertenprogramm des Cells in Motion Interfaculty Centres der WWU. Um die dreidimensionalen Daten mehrerer Embryonen digital „übereinanderlegen“ zu können, taten sich die Wissenschaftler aus der Biologie mit einem Team um den Mathematiker Prof. Dr. Martin Burger zusammen, der damals ebenfalls an der WWU forschte und die Zusammenarbeit heute an der FAU fortsetzt. Das Team entwickelte eine neue Software, die es ermöglicht, Mikroskopiedatensätze automatisiert zusammenzufügen und Muster in der Verteilung der Urkeimzellen zu erkennen. Die Herausforderung: Die einzelnen Zebrafischembryonen unterscheiden sich in ihrer Größe und Form, und die Mikroskopieaufnahmen weichen in ihrer dreidimensionalen Ausrichtung voneinander ab.
Die Software mit dem Namen „Landscape“ bringt die Bilder aller aufgenommenen Embryonen in eine Übereinstimmung miteinander. „Auf Grundlage einer Segmentierung der Zellkerne analysiert die Software zunächst die Größe und Form der Embryonen und gleicht diese an. Anschließend erfolgt eine Korrektur der Körperausrichtung“, erklärt der Mathematiker Dr. Daniel Tenbrinck, ebenfalls Erstautor der Studie. Dabei dient die Längsachse der Embryonen als Referenzstruktur, die durch eine gewebespezifische Expression des sogenannten grün fluoreszierenden Proteins (GFP) markiert ist. Im Fachjargon nennt sich der ganze Vorgang Bildregistrierung. Die Wissenschaftler überprüften die Zuverlässigkeit ihrer Algorithmen, indem sie mehrere Bilder desselben Embryos aufnahmen, diese hinsichtlich Größe und Bildorientierung manipulierten, und das Ergebnis der Bildregistrierung mit dem Originalbild verglichen. Um ihre Methodik zu evaluieren, nutzten sie zudem Mikroskopiebilder von sich normal entwickelnden Embryonen, von denen bekannt ist, an welchen Stellen sich Urkeimzellen anhäufen. Die Forscher zeigten auch, dass sich die Software bei Embryonen eines anderen Modellorganismus anwenden lässt – der Taufliege Drosophila – die eine länglichere Form als Zebrafischembryonen haben.
Mit der neuen Methode analysierten die Wissenschaftler die Verteilung von rund 21.000 Urkeimzellen in 900 Zebrafischembryonen. Wie erwartet ordneten sich die Zellen ohne Chemokinrezeptor anders an als sonst. Dabei zeigten sie ein eindeutiges Verteilungsmuster, das in der Untersuchung einzelner Embryonen nicht zu erkennen war. So blieb die Region um die Längsachse der Embryonen fast vollständig frei von Zellen. Bei einer genaueren Betrachtung fanden die Forscher heraus, dass diese Region als physikalische Barriere fungiert: Bei Kontakt mit der Gewebebarriere verändern die Zellen die Verteilung des Proteins Aktin, was zu einer Richtungsänderung ihrer Fortbewegung weg von der Barriere führt. Ein tieferes Verständnis darüber, wie Zellen auf physikalische Barrieren reagieren, könnte bei metastasierenden Krebszellen relevant sein, die in Nachbargewebe eindringen und bei denen dieser Prozess gestört sein könnte.
Förderung
Die Studie erhielt finanzielle Unterstützung durch die Deutsche Forschungsgemeinschaft, insbesondere im Rahmen des Exzellenzclusters „Cells in Motion“ (2012-2019) und des Sonderforschungsbereichs 1348 „Dynamische zelluläre Grenzflächen: Bildung und Funktion“ der WWU. Zudem trugen Fördermittel des Cells in Motion Interfaculty Centres und der Medizinischen Fakultät der WWU, des European Molecular Biology Laboratory, des Bundesministeriums für Bildung und Forschung sowie der Europäische Union (MSCA-RISE „Nonlocal Methods for Arbitrary Data Sources“, NoMADS) zur Umsetzung der Studie bei.
Originalpublikation
Gross-Thebing S, Truszkowski L, Tenbrinck D, Sánchez-Iranzo H, Camelo C, Westerich KJ, Singh A, Maier P, Prengel J, Lange P, Hüwel J, Gaede F, Sasse R, Vos BE, Betz T, Matis M, Prevedel R, Luschnig S, Diz-Muñoz A, Burger M, Raz E. Using migrating cells as probes to illuminate features in live embryonic tissues. Sci Adv 2020;6: eabc5546.
